Ⅰ. 반응 시스템의 감도를 높입니다.
1. 고품질 RNA 분리:
성공적인 cDNA 합성은 고품질 RNA에서 나옵니다.고품질 RNA는 적어도 총 길이가 더 길어야 하며 EDTA 또는 SDS와 같은 기록 효소를 포함하지 않는 억제제를 포함하지 않습니다.RNA의 품질은 cDNA에 전사할 수 있는 서열 정보의 최대값을 결정합니다.일반적인 RNA 정제 방법은 isoocyanate/acidophenol을 이용하는 단계적 방법이다.RNase의 오염을 방지하기 위해 RNase가 풍부한 샘플(예: 췌장)에서 분리된 RNA는 고품질 RNA를 저장하기 위해 포름알데히드 저장이 필요하며, 장기 저장의 경우 더욱 그렇습니다.쥐의 간에서 추출한 RNA는 물에서 1주일 보관하면 기본적으로 분해되는 반면, 쥐의 비장에서 추출한 RNA는 물에서 3년 보관해도 안정한 상태를 유지했다.또한 4kb보다 큰 전사체는 작은 전사체보다 추적 RNase 분해에 더 민감합니다.보관 RNA 샘플의 안정성을 높이기 위해 RNA를 이온의 메탈아민에 용해하여 -70°C에 보관할 수 있습니다.RNA를 저장하는 데 사용되는 Thylide는 RNA를 분해하는 잡다한 물체를 포함하지 않아야 합니다.췌장에서 유래한 RNA는 적어도 1년 동안 methalmamine에 저장할 수 있습니다.RNA를 사용할 준비가 되면 다음 방법을 사용하여 RNA를 침전시킬 수 있습니다: 0.2m에 NaCl을 첨가하고 에탄올 부피의 4배, 실온에 3-5분 동안 두고 10,000 × g에서 5분 동안 원심분리합니다.
2. RNaseH 활성이 없는 역전사 효소(RNaseH-) 사용:
RNase 억제제는 종종 역전사 반응에 첨가되어 cDNA 합성의 길이와 수율을 증가시킵니다.RNase 억제제는 pre-cDNA 합성 과정이 억제제를 변성시켜 RNA를 분해하는 결합된 RNase를 방출하기 때문에 DTT와 같은 완충제 및 환원제가 있는 상태에서 첫 번째 연쇄 합성 반응에 추가됩니다.Protein RNase inhibitor는 RNase A, B, C에 의한 RNA의 분해만 방지할 뿐, 피부에 RNases를 방지하지 못하므로 이러한 억제제를 사용함에도 불구하고 손가락에서 RNases가 유입되지 않도록 주의해야 합니다.
역전사 효소는 RNA를 cDNA로 전환하는 것을 촉매합니다.M-MLV와 AMV는 둘 다 그들 자신의 중합효소 활성에 더하여 내인성 RNaseH 활성을 가지고 있습니다.RNaseH 활성은 RNA 주형과 DNA 프라이머 또는 cDNA 연장 가닥 사이에 형성된 이형접합 가닥에 대해 중합효소 활성과 경쟁하며, RNA를 분해합니다: DNA 복합체의 RNA 가닥.RNaseH 활성에 의해 분해된 RNA 템플릿은 더 이상 cDNA 합성을 위한 효과적인 기질로 사용할 수 없으므로 cDNA 합성의 수율과 길이가 감소합니다.따라서 역전사효소의 RNaseH 활성을 제거하거나 크게 줄이는 것이 큰 이점이 될 것입니다.
RNaseH-의 SuperScriptⅡ 역전사효소, MMLV 역전사효소 및 RNaseH-의 thermoScript 역전사효소 AMV는 MMLV 및 AMV보다 더 많은 전장 cDNA를 생성하였다.RT-PCR 민감도는 합성된 cDNA의 양에 영향을 받습니다.ThermoScript는 AMV보다 훨씬 더 민감합니다.RT-PCR 제품의 크기는 특히 더 큰 Cdnas를 복제할 때 cDNA를 합성하는 역전사 효소의 능력에 의해 제한됩니다.MMLV와 비교하여 SuperScripⅡ는 긴 RT-PCR 제품의 수율을 크게 높였습니다.RNaseH-의 역전사효소도 열적 안정성을 높여주기 때문에 정상보다 높은 온도인 37~42℃에서도 반응이 가능하다.제안된 합성 조건에서 oligo(dT) 프라이머와 10μCi [alpha-p]dCTP를 사용하였다.첫 번째 체인의 총 생산량은 TCA 침전 방법을 사용하여 계산되었습니다.전체 길이 cDNA는 크기별로 분류된 스트립 제거 및 알칼리성 아가로스 겔에서 계수를 사용하여 분석되었습니다.
3. 역전사의 보온 온도를 높이십시오.
더 높은 유지 온도는 RNA의 2차 구조를 열고 반응 수율을 높이는 데 도움이 됩니다.대부분의 RNA 템플릿의 경우 RNA와 프라이머를 완충액이나 염 없이 65°C에서 유지한 다음 얼음 위에서 빠르게 냉각하면 대부분의 2차 구조가 제거되고 프라이머가 결합할 수 있습니다.그러나 일부 템플릿은 열 변성 후에도 여전히 2차 구조를 가지고 있습니다.이러한 어려운 주형의 증폭은 ThermoScript 역전사 효소를 사용하고 역전사 효소 반응을 더 높은 온도에서 수행하여 증폭을 개선할 수 있습니다.유지 온도가 높을수록 특히 유전자 특이 프라이머(GSPS)를 사용하여 cDNA 합성을 수행할 때 특이성이 증가할 수 있습니다(3장 참조).GSP를 사용하는 경우 프라이머의 Tm 값이 예상 유지 온도와 동일한지 확인하십시오.60℃ 이상의 올리고(dT) 및 random primer는 사용하지 마십시오.랜덤 프라이머는 60℃로 올리기 전에 10분 동안 25℃에서 유지해야 합니다.더 높은 역전사 온도를 사용하는 것 외에도 RNA/프라이머 혼합물을 65℃ 변성 온도에서 역전사 유지 온도로 직접 옮기고 예열된 2X 반응 혼합물을 추가하여 특이성을 향상시킬 수 있습니다(cDNA 열 개시 합성).이 접근법은 낮은 온도에서 발생하는 분자간 염기쌍 형성을 방지하는 데 도움이 됩니다.PCR 장비를 사용하면 RT-PCR에 필요한 많은 온도 스위치를 단순화할 수 있습니다.
열 안정화 중합효소는 Mg2+ 존재 시 DNA 중합효소로 작용하고 Mn2+ 존재 시 RNA 중합효소로 작용합니다.65℃까지 열을 유지할 수 있습니다.그러나 PCR 동안 Mn2+의 존재는 충실도를 감소시켜 Tth 중합효소가 cDNA 클로닝과 같은 고정밀 증폭에 적합하지 않게 만듭니다.또한 Tth는 역전사 효율이 낮아 감도가 떨어지고 단일 효소가 역전사 및 PCR을 수행할 수 있기 때문에 역전사 없는 제어 반응으로는 cDNA 증폭 산물과 오염된 genomic DNA 증폭 산물을 구별할 수 없습니다.
4. 역전사 촉진 첨가제:
글리세린과 DMSO를 포함한 첨가제를 1차 연쇄 합성 반응에 첨가하면 핵산 이중 가닥의 안정성을 감소시키고 RNA 2차 구조를 풀 수 있습니다.SuperScriptⅡ 또는 MMLV의 활동에 영향을 주지 않고 최대 20% 글리세린 또는 10% DMSO를 추가할 수 있습니다.AMV는 또한 활성 감소 없이 최대 20%의 글리세롤을 견딜 수 있습니다.SuperScriptⅡ 역전사 반응에서 RT-PCR의 감도를 극대화하기 위해 10% 글리세롤을 첨가하여 45℃에서 절연할 수 있습니다.역전사 반응 산물의 1/10을 PCR에 첨가하면 증폭 반응의 글리세롤 농도는 0.4%로 PCR을 억제하기에 충분하지 않다.
5. RNaseH 처리:
PCR 전에 cDNA 합성 반응을 RNaseH로 처리하여 감도를 향상시킬 수 있습니다.일부 템플릿의 경우 cDNA 합성 반응의 RNA가 증폭된 산물의 결합을 방지하는 것으로 생각되며, 이 경우 RNaseH 처리가 감도를 높일 수 있습니다.일반적으로 RNaseH 처리는 copy가 적은 tuberous scherosisⅡ와 같이 상대적으로 긴 full-length cDNA target template의 증폭을 위해 필요합니다.이 어려운 템플릿의 경우 RNaseH는 SuperScriptⅡ 또는 AMV로 합성된 cDNA에서 생성된 신호를 강화했습니다.대부분의 RT-PCR 반응에서 95℃ 절연 PCR 변성 단계는 일반적으로 RNA:DNA 복합체에서 RNA를 가수분해하기 때문에 RNaseH 처리는 선택 사항입니다.
6. 소량의 RNA 검출을 위한 개선된 방법:
RT-PCR은 소량의 RNA만 사용할 수 있을 때 특히 어렵습니다.RNA 분리 중 글리코겐을 캐리어로 추가하면 작은 샘플의 수율을 높이는 데 도움이 됩니다.RNase-free 글리코겐은 Trizol과 동시에 추가할 수 있습니다.글리코겐은 수용성이며 후속 침전을 돕기 위해 RNA와 함께 수상에 남아 있을 수 있습니다.RNase-free 글리코겐의 권장 농도는 50mg 미만의 조직 또는 106개 배양 세포 샘플의 경우 250μg/ml입니다.
SuperScriptⅡ를 이용한 역전사 반응에 acetylated BSA를 첨가하면 민감도를 높일 수 있으며 소량의 RNA에 대해서는 SuperScriptⅡ의 양을 줄이고 RnaseOut nuclease inhibitor를 40unit 첨가하면 검출율을 높일 수 있다.RNA 분리에 글리코겐을 사용하는 경우 SuperScriptⅡ를 사용하여 역전사 반응에 BSA 또는 RNase 억제제를 추가하는 것이 여전히 권장됩니다.
Ⅱ. RT-PCR의 특이성을 높입니다.
1. cNDA 합성:
세 가지 다른 방법을 사용하여 첫 번째 가닥 cDNA 합성을 시작할 수 있으며 각 방법의 상대적 특이성은 합성되는 cDNA의 양과 유형에 영향을 미칩니다.
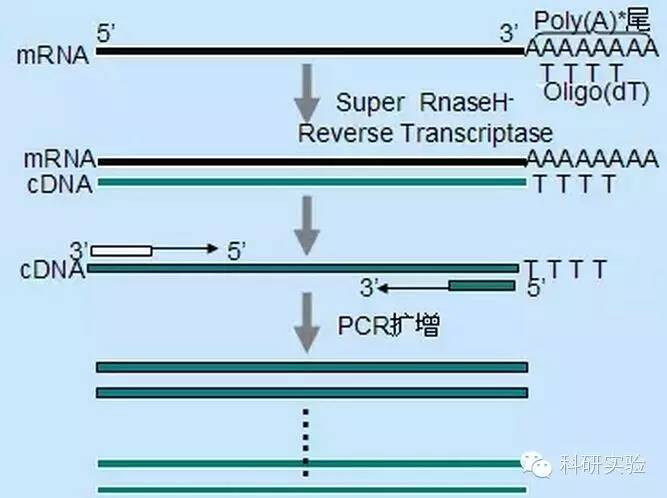
무작위 프라이머 방법은 세 가지 방법 중 가장 특이성이 낮습니다.프라이머는 짧은 부분 길이의 cDNA를 생성하기 위해 전사체 전체에 걸쳐 여러 위치에서 어닐링됩니다.이 방법은 종종 역전사 효소가 복제할 수 없는 2차 구조 영역 또는 종결 부위가 있는 RNA 주형으로부터 5' 말단 서열 및 cDNA를 얻는 데 사용됩니다.가장 긴 cDNA를 얻으려면 각 RNA 샘플에서 RNA에 대한 프라이머의 비율을 경험적으로 결정해야 합니다.랜덤 프라이머의 초기 농도는 20μl 반응 시스템당 50~250ng 범위입니다.random primer를 이용하여 total RNA로부터 합성한 cDNA는 주로 ribosomal RNA이기 때문에 일반적으로 poly(A)+RNA를 template로 선택합니다.
Oligo(dT) 개시는 랜덤 프라이머보다 더 특이합니다.이것은 대부분의 진핵 세포에서 mRNA의 3' 말단에서 발견되는 폴리(A) 꼬리와 혼성화합니다.poly(A)+RNA는 총 RNA의 약 1%~2%이기 때문에 cDNA의 양과 복잡성은 무작위 프라이머를 사용하는 경우보다 훨씬 적습니다.특이성이 높기 때문에 oligo(dT)는 일반적으로 RNA 대 프라이머 비율 및 poly(A)+ 선택에 대한 최적화가 필요하지 않습니다.20μl reaction system당 0.5μg oligo(dT) 사용을 권장합니다.oligo(dT)12-18은 대부분의 RT-PCR에 적합합니다.ThermoScript RT-PCR System은 우수한 열 안정성으로 인해 oligo(dT)20을 제공하며 높은 유지 온도에 적합합니다.
유전자 특이적 프라이머(GSP)는 역전사 단계에 가장 적합한 특이적 프라이머입니다.GSP는 랜덤 프라이머 또는 올리고(dT)와 같은 모든 Rnas를 어닐링하기보다는 RNA 대상 서열과 특이적으로 혼성화할 수 있는 안티센스 올리고뉴클레오사이드입니다.PCR 프라이머 설계에 사용되는 규칙은 역전사 반응 GSP 설계에도 적용됩니다.GSP는 mRNA3' 말단에서 어닐링된 증폭 프라이머와 동일한 서열일 수 있거나, GSP는 역 증폭 프라이머로 하류에서 어닐링되도록 설계될 수 있습니다.일부 증폭된 개체의 경우 표적 RNA의 2차 구조로 인해 프라이머가 결합하지 못할 수 있으므로 성공적인 RT-PCR을 위해 하나 이상의 안티센스 프라이머를 설계해야 합니다.20μl의 1차 연쇄 합성 반응 시스템에서 1pmol 안티센스 GSP를 사용하는 것이 좋습니다.
2. 역전사의 보온 온도를 높이십시오.
GSP 특이성을 최대한 활용하기 위해서는 열 안정성이 높은 역전사 효소를 사용해야 합니다.열에 안정한 역전사효소는 더 높은 온도에서 격리되어 반응 엄격성을 높일 수 있습니다.예를 들어, GSP가 55°C에서 어닐링되는 경우 AMV 또는 M-MLV를 사용하여 낮은 엄격도로 37°C에서 역전사를 수행하면 GSP의 특이성이 완전히 활용되지 않습니다.그러나 SuperScripⅡ와 ThermoScript는 50℃ 이상에서 반응할 수 있어 더 낮은 온도에서 생성되는 불특정한 제품을 제거합니다.최대 특이성을 위해 RNA/프라이머 혼합물은 예열된 2x 반응 혼합물(cDNA 합성의 열 개시)을 추가하여 65℃ 변성 온도에서 역전사 유지 온도로 직접 옮길 수 있습니다.이는 저온에서 분자 사이의 염기쌍 형성을 방지하는 데 도움이 됩니다.PCR 기기를 사용하면 RT-PCR에 필요한 많은 온도 전환이 간소화됩니다.
3. 게놈 DNA 오염 감소:
RT-PCR의 한 가지 잠재적인 어려움은 RNA가 게놈 DNA를 오염시킨다는 것입니다.Trizol Reagent와 같은 더 나은 RNA 분리 방법을 사용하면 RNA 준비에서 게놈 DNA 오염이 줄어듭니다.게놈 DNA에서 생산된 제품을 피하기 위해 RNA는 역전사 전에 오염된 DNA를 제거하기 위해 증폭 등급 DnasⅠ로 처리될 수 있습니다.샘플을 DNaseⅠ 소화를 종료하기 위해 10분 동안 2.0mM EDTA에서 65℃로 유지하였다.EDTA는 마그네슘 이온을 킬레이트화하여 고온에서 발생하는 마그네슘 이온 의존성 RNA 가수분해를 방지합니다.
게놈 DNA 증폭 산물에서 증폭된 cDNA를 분리하기 위해서는 분리된 엑손과 별도로 어닐링하는 프라이머를 설계할 수 있다.cDNA에서 파생된 PCR 제품은 오염된 게놈 DNA에서 파생된 제품보다 짧습니다.주어진 단편이 게놈 DNA 또는 cDNA에서 온 것인지를 결정하기 위해 각 RNA 주형에서 역전사 없이 통제된 실험도 수행됩니다.역전사 없이 얻은 PCR 산물은 게놈에서 파생됩니다.
관련 제품
-One-step kit로 역전사와 PCR이 같은 tube에서 가능합니다.템플릿 RNA, 특정 PCR 프라이머 및 RNase-Free ddH만 추가하면 됩니다.2O.
-RNA의 실시간 정량 분석을 빠르고 정확하게 수행할 수 있습니다.
-독특한 Foregene 역전사 시약과 Foregene HotStar Taq DNA Polymerase를 독자적인 반응 시스템과 결합하여 반응의 증폭 효율과 특이성을 효과적으로 향상시키는 키트입니다.
-최적화된 반응 시스템은 반응이 더 높은 검출 감도, 더 강한 열 안정성 및 더 나은 내성을 갖도록 합니다.
RT Easy II(With GDNase) Master Premix for First-Strand CDNA Synthesis For Real Time PCR With GDNase
-gDNA를 효율적으로 제거하는 능력으로 template내의 gDNA를 2분 이내에 제거할 수 있습니다.
-효율적인 역전사 시스템으로 첫 번째 가닥 cDNA의 합성을 완료하는 데 15분밖에 걸리지 않습니다.
-복잡한 템플릿: GC 함량이 높고 복잡한 2차 구조가 있는 템플릿도 높은 효율로 역전할 수 있습니다.
- 고감도 역전사 시스템, pg 수준의 템플릿으로도 고품질의 cDNA를 얻을 수 있습니다.
-역전사 시스템은 열적 안정성이 높고, 최적의 반응 온도는 42℃이며, 50℃에서도 역전사 성능이 여전히 양호합니다.
게시 시간: 2023년 3월 7일